Aguas negras: un ecosistema complejo que nos ayuda a anticipar epidemias
Rodrigo García López
El Dr. García López estudió la Licenciatura en Ciencias Genómicas en la UNAM (Cuernavaca, Morelos). Obtuvo la Maestría en Biología Molecular, Celular y Genética y el Doctorado en Biotecnología en la Universidad de Valencia (España). Es investigador en el Instituto de Biotecnología de la UNAM, encabezando una línea de investigación sobre monitoreo de virus en aguas residuales. Es Investigador Nacional Nivel 1 e integrante de la Academia de Ciencias de Morelos.
Esta publicación fue revisada por el comité editorial de la Academia de Ciencias de Morelos.
La mayoría consideramos las aguas negras tan solo como un subproducto de desecho de la población, contaminación humana que termina vertida en ríos y mares o, en el mejor de los casos, tratada para su reutilización en riego u otros fines. Sin embargo, las aguas residuales son también un valioso recurso para estudiar enfermedades humanas, anticiparse a brotes, identificar patógenos resistentes o servir para estimar el abuso de sustancias.
De pandemia en pandemia
Comenzamos la década de los años 20s de este siglo con la pandemia de COVID-19, causada por el virus SARS-CoV-2. Sin duda, esta fue una emergencia sanitaria global que vino a trastocar de forma irreversible las vidas de la gran mayoría de las personas en el planeta. Sin embargo, ésta llegó a México tan sólo una década después de otra pandemia previa: aquella causada por el virus Influenza A subtipo H1N1 en 2009. Esta pandemia fue conocida popularmente como la gripe porcina, que además es recordada por haber tenido su origen en nuestro país (1). Ambos virus causaron crisis importantes y alarma al inicio de sus respectivas pandemias, pero la evolución los llevó a adaptarse a la población humana conforme esta, a su vez, adquiría inmunidad parcial. Como resultado, tanto la COVID-19 como la influenza pandémica H1N1 permanecen en la población como enfermedades estacionales, representando un riesgo principalmente para determinados grupos vulnerables, como la infancia y los adultos mayores. Las tristes consecuencias de ambas pandemias, causadas por dos virus muy diferentes, nos dejan como lecciones la importancia de prepararnos para nuevos eventos epidémicos, y mantener o mejorar la infraestructura para monitoreo de enfermedades.
Tras la declaratoria del fin de la fase de emergencia de la pandemia de COVID-19 por parte de la Organización Mundial de la Salud en mayo de 2023 (9), la mayoría de los países se prepararon para reducir el esfuerzo del sistema de vigilancia que hasta entonces había permitido conocer el avance regional del virus SARS-CoV-2 y sus variantes dominantes. Esto era razonable, porque el virus había terminado por volverse endémico y la mayoría en la población contaba ahora con cierto grado de protección inmunológica. Esta protección llegó gracias a la aplicación de la vacuna y sus refuerzos, o por infecciones durante la pandemia (la mayoría siendo pacientes con enfermedad leve o asintomática). En México, nos llegó el turno para la desescalada de la vigilancia, comenzando en septiembre de 2023, momento a partir del cual oficialmente dejan de contabilizarse pruebas que no sean confirmadas por la reacción en cadena de la polimerasa o PCR (Figura 1). La PCR es sin duda la técnica más confiable, pero representa menos del 5% de todas las pruebas realizadas diariamente a nivel nacional; desde 2021, la gran mayoría de la pandemia se monitoreó con pruebas rápidas de antígeno (2).
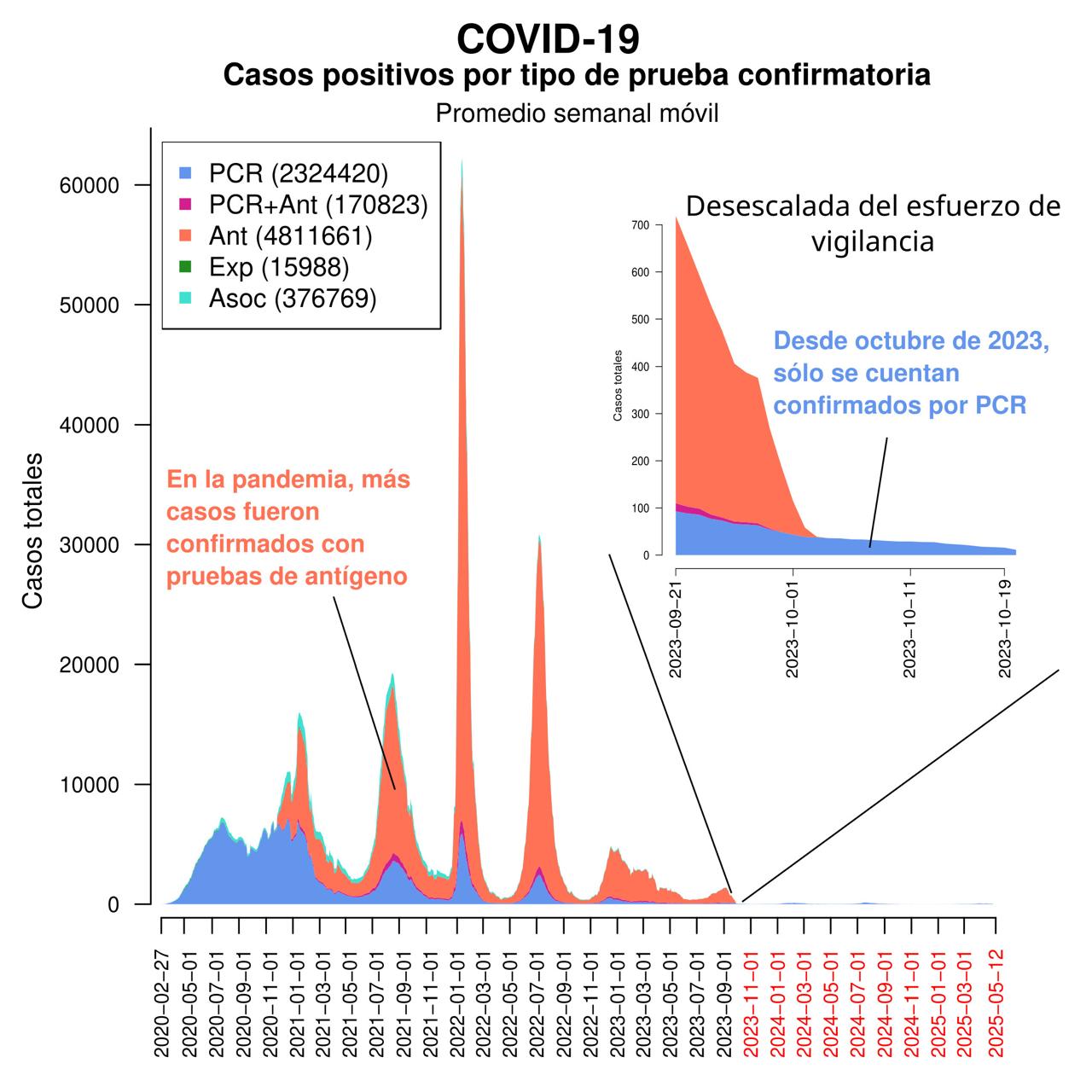
Figura 1. Resumen histórico de casos positivos de COVID-19 en México, por tipo de prueba confirmatoria. Se muestra el detalle de septiembre-octubre de 2023, cuando se dejaron de contar las pruebas de antígeno en los datos oficiales. PCR: Prueba por Reacción en Cadena de la Polimerasa; Ant: Prueba rápida de antígeno; Exp: Por dictaminación experta; Asoc: Por asociación. Gráfica elaborada a partir de los datos abiertos de la Dirección General de Epidemiología actualizados a 13 de mayo de 2025.
A raíz del cambio de protocolo de vigilancia, la COVID-19, al igual que el resto de enfermedades respiratorias en México, ha pasado a ser monitoreada de forma reactiva, ya no en tiempo real (como es lógico, al no representar ya una emergencia). Si bien los protocolos oficiales de la Secretaría de Salud garantizan el monitoreo centinela de todas las principales poblaciones del país, ésta vigilancia clínica tiene algunas limitaciones. Actualmente considera sólo a pacientes que se atienden en el sistema de salud público (durante pandemia participaron los privados). Adicionalmente, se concentra en pacientes cuya infección los lleva a atenderse en un hospital o clínica (de los asintomáticos sabemos más bien poco). Como resultado, el panorama epidemiológico actual que tenemos de las enfermedades respiratorias es bastante parcial en el país. Para darse una idea de la magnitud de este problema, mediante indicadores menos sesgados por la falta de muestras, tales como la positividad (de entre todos los casos analizados, cuántos resultan positivos), se puede observar que el virus SARS-CoV-2 habría causado nuevas olas durante 2024 y 2025, al igual que en años anteriores (Figura 2). Sin embargo, el escaso número de muestras es insuficiente para estudiar a detalle la situación real.
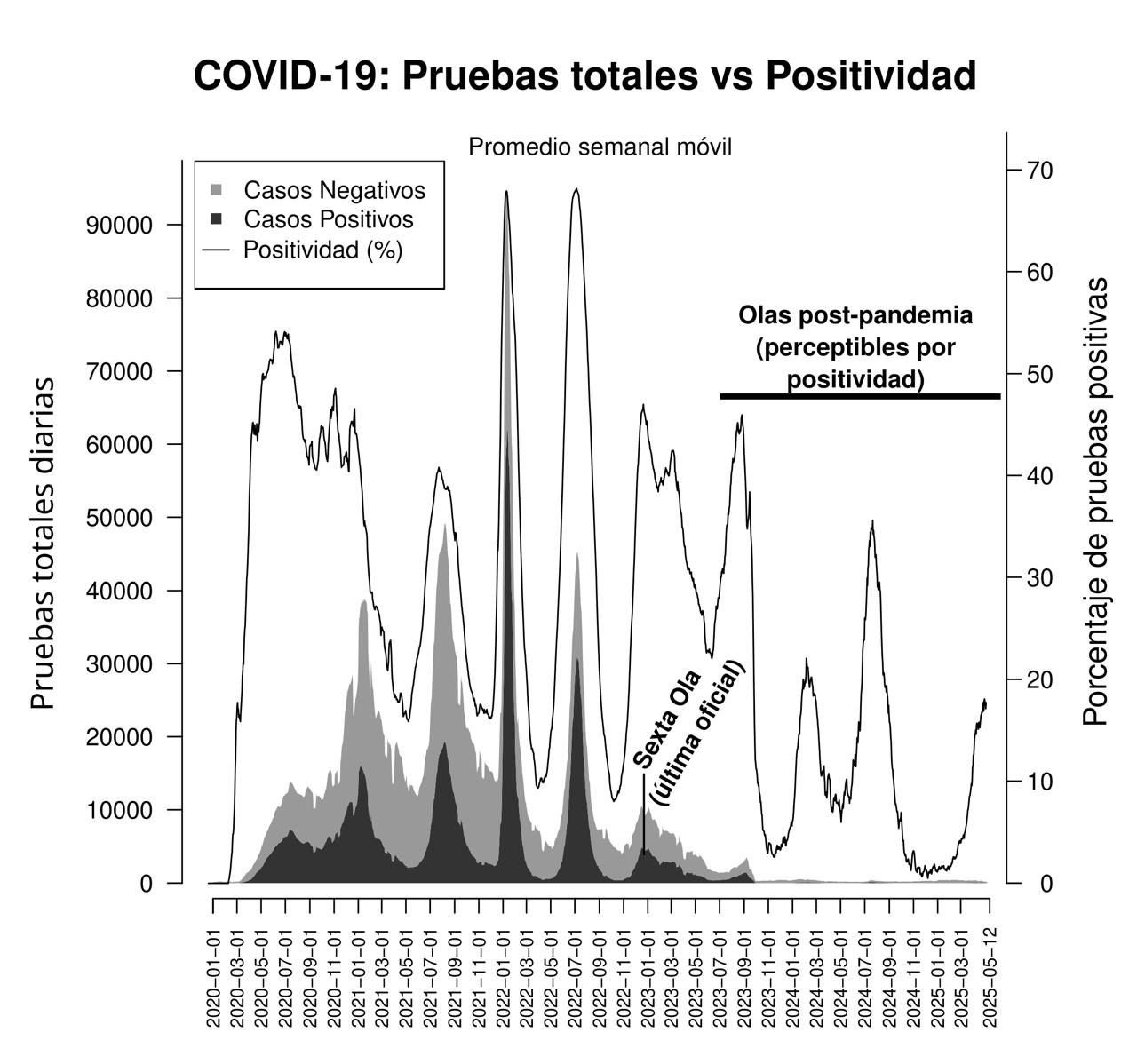
Figura 2. Registro histórico en México de pruebas positivas y negativas desde el inicio de la pandemia de COVID-19 con positividad. Las últimas cuatro subidas epidemiológicas del virus se pueden observar únicamente a través de la positividad (porcentaje de pruebas que resultan positivas). Gráfica elaborada a partir de los datos abiertos de la Dirección General de Epidemiología actualizados a 13 de mayo de 2025.
Virus y bacterias en nuestro drenaje
Ante la nueva realidad postpandemia y debido a la disminución de muestras clínicas, diversos países del norte global (antes llamados “desarrollados”) han optado por implementar e institucionalizar un sistema de monitoreo diferente para seguir las enfermedades. Este es un esfuerzo complementario e independiente de la vigilancia clínica, pero capaz de detectar de forma anticipada los brotes epidémicos: un sistema de Vigilancia de Aguas Residuales (VAR). De manera general, la VAR consiste en tomar muestras de aguas negras para evaluar la presencia y concentración de patógenos, como virus o bacterias, o contaminantes químicos, que proceden de una población. Así, una muestra de agua tomada de una alcantarilla o planta de tratamiento da una idea de los posibles impactos a la salud para cientos o miles de personas, un monitoreo mucho más amplio de lo que puede decirnos un único paciente (10).
¿Pero qué es lo que buscamos en el agua residual? El intestino humano, además de ser clave para la absorción de los nutrientes en nuestros alimentos, es un lugar rico en vida pues también es el hogar y lugar de tránsito para millones de organismos microscópicos, incluidas cientos de especies de bacterias, hongos, parásitos unicelulares y partículas virales que, en conjunto, forman lo que conocemos como microbioma humano. La gran mayoría de estos organismos no son dañinos, sino que forman parte de una compleja red de interacción ecológica natural que tiene un impacto en los procesos digestivos, metabólicos y de inmunidad que contribuyen a la salud humana. Estos organismos son excretados en las heces, orina, descamación de la piel y otros fluidos, hacia el drenaje y son un marcador de qué tan sana está una población o, cuando hay patógenos, de cómo avanza un brote epidémico (4). La abundancia de estos microoganismos es lo que se busca al hacer VAR, y se hace a través de la detección de su material genético.
Estudios de las aguas negras, más de 170 años salvando vidas
La primera implementación estandarizada de la vigilancia de enfermedades de una población a través de sus aguas residuales se le atribuye al Dr. John Snow. Este médico británico estudió por primera vez un brote de cólera ocurrido en Londres en 1854. Su cuidadoso estudio, documentando en mapas el avance de la enfermedad, le permitió identificar la fuente de la infección a tomas de agua contaminadas con materia fecal (7). Este trabajo marca el nacimiento de la epidemiología, disciplina de la salud pública que estudia el origen, distribución, frecuencia y magnitud de los brotes epidémicos. Durante los 1920s, grupos británicos como el de James Wilson en Gales, lograron estandarizar los primeros protocolos de microbiología para hacer vigilancia de bacterias patógenas (causantes de enfermedades) en las aguas residuales (8), logrando cultivar bacterias entéricas (actualmente identificadas como Salmonella enterica subtipos typhi o paratyphi) para dar seguimiento a brotes de fiebre tifoidea. Quedó de manifiesto que se podía lograr la detección de los patógenos directamente del agua de desecho de una comunidad, aún en ausencia de casos clínicos reportados, una ventaja que permite estudiar una enfermedad que se transmite de manera silenciosa en una población (asintomática en la mayor parte de individuos).
El siguiente gran impulso hacia la adopción de la VAR como herramienta de monitoreo llegó hacia el final de los años 1930s y comienzo de los 1940s, cuando John Paul y James Trask emplearon el estudio de las aguas residuales para monitorear el avance de la poliomielitis en Filadelfia (5). Debido a que sólo 1 de cada 200 casos de esta enfermedad resulta paralizante, los brotes de Poliovirus pueden pasar desapercibidos hasta que aparecen los primeros casos, momento en el cual la enfermedad ya se ha diseminado en la población. La VAR se presentó como una alternativa atractiva a la epidemiología tradicional clínica, pues permitía confirmar la presencia de la enfermedad con antelación suficiente para aplicar medidas de prevención en una población. Tras el desarrollo y aplicación de las primeras vacunas contra la poliomielitis en las dos décadas siguientes, la VAR mediante cultivo de virus comenzó a adoptarse internacionalmente para evaluar la remisión del virus en las poblaciones humanas. Terminaría por consolidarse como una como herramienta imprescindible para la epidemiología durante las campañas de erradicación del virus (1990 fue el último caso de virus silvestre de transmisión local), cuando la OMS recomendó la actualización de la VAR con técnicas moleculares no dependientes de cultivo y con más fino umbral de detección (como la PCR y derivadas) para medir su avance.
En México, la adopción de este tipo de vigilancia ha sido tardía, discreta y regional, dando inicio en la década de 2010 con seguimientos puntuales en áreas urbanas de las cepas vacunales de polio antes del retiro de la vacuna oral en 2020 (3). Mediante VAR se confirmó que cepas de poliovirus atenuadas, derivadas de la vacuna oral, continuaban circulando durante algunos meses incluso en poblaciones con poca vacunación (algo que ya no sucede con las vacunas actuales). Durante la pandemia de COVID-19, las técnicas para VAR se actualizaron en México para hacer monitoreo del virus SARS-CoV-2, logrando su detección en cuerpos de agua contaminados como ríos (11) y plantas de tratamiento urbanas (6) pero no trascendió más allá del interés académico. Pese al creciente interés y aunque existen nuevas iniciativas para llevar a cabo VAR en distintas regiones del país, continúa siendo un esfuerzo desarticulado, con retos pendientes de resolver en cuanto a infraestructura, logística y vinculación con el sector salud.
VAR en Morelos
Desde 2024, el grupo de la Dra. Luz Bretón Deval y el Dr. Rodrigo García López en el Instituto de Biotecnología (IBt) de la UNAM en Cuernavaca, Morelos, comenzó a trabajar en el desarrollo de una plataforma de monitoreo a partir de aguas residuales que sea compatible con nuestra realidad en México, enfocada en las bacterias y los virus de poblaciones urbanas. El enfoque emplea técnicas de metagenómica, las que permiten, sin necesidad de cultivos, analizar el contenido completo de material genético en una muestra. Por tanto,no se limita a la detección de organismos patógenos individuales sino que abarca el espectro completo de bacterias y virus ambientales que se alojan en las muestras de aguas negras. Esto nos permitirá entender el papel ecológico de esos microorganismos, sus adaptaciones evolutivas y la posible presencia y variedad de genes de resistencia a antibióticos en ellos.
Actualmente, el grupo desarrolla un proyecto piloto con muestras de la planta de tratamiento de aguas “El Rayo” en la población de Temixco. Esta planta procesa aguas negras para regresarlas al río Apatlaco (Figura 3), dando servicio a la mayor parte de la población del municipio (alrededor de 85,000 habitantes). La cisterna de entrada de la planta sirve como concentrador del flujo de las aguas negras proveniente de miles de hogares y negocios, haciendo que estudiar las muestras de la planta sea una manera rápida y eficiente de revisar aquello que circula en la población completa, de una sola vez. Por otro lado, se busca que la muestra no sea una toma de agua sencilla (que en una analogía sería como emplear una fotografía de un momento único en el día), sino un volumen continuo recolectado a lo largo del 24 horas (en la misma analogía, sería el equivalente a una película) por lo que el grupo del IBt ha desarrollado un prototipo de dispositivo de muestreo que es fácilmente reproducible y aplicable en otras plantas.

Figura 3. Toma de muestra en la planta de tratamiento “El Rayo” en Temixco. A, Vista general de la planta; B, Río Apatlaco en su paso por Temixco; C, Cisterna de entrada de la planta; D, Primeras pruebas del dispositivo de muestreo.
En su etapa inicial, el seguimiento epidemiológico en Temixco se enfoca en bacterias patógenas comunes, como algunas cepas de Klebsiella y Pseudomonas, así como en los virus estacionales SARS-CoV-2 (COVID-19) e Influenza A (gripe), pero es tan sólo la primera etapa de un esfuerzo que continuará creciendo para incluir más sitios de Morelos así como un universo mucho más amplio de microorganismos mediante metagenómica. Se espera lograr contribuir en establecer en Morelos un mecanismo de vigilancia epidemiológica estable que sea complementario al seguimiento que hace el sistema de salud para detectar de forma oportuna cambios en la proporción de microorganismos en la población.
Referencias:
- Cordova-Villalobos JA, Macias AE, Hernandez-Avila M, Dominguez-Cherit G, Lopez-Gatell H, Alpuche-Aranda C, Ponce de León-Rosales S. (2017). The 2009 pandemic in Mexico: Experience and lessons regarding national preparedness policies for seasonal and epidemic influenza. Gac Med Mex. 153(1):102-110. https://www.anmm.org.mx/GMM/2017/n1/GMM_153_2017_1_102-110.pdf
- Dirección General de Epidemiología (DGE). Lineamiento estandarizado para la vigilancia epidemiológica y por laboratorio de la enfermedad respiratoria viral. 2023. Consultada el 24 de febrero de 2025 en https://www.gob.mx/cms/uploads/attachment/file/636504/Lineamiento_Estandarizado_Para_La_Vigilancia_Epidemiol_gica_Y_Por_Laboratorio_De_La_Enfermedad_Respiratoria_Viral_-_Septiembre_2023.pdf
- Esteves-Jaramillo A, Estívariz CF, Peñaranda S, Richardson VL, Reyna J, Coronel DL, Carrión V, Landaverde JM, Wassilak SG, Pérez-Sánchez EE, López-Martínez I, Burns CC, Pallansch MA (2014). Detection of vaccine-derived polioviruses in Mexico using environmental surveillance. J Infect Dis. 210 (Suppl 1):S315-323. doi: 10.1093/infdis/jiu183.
- Parkins MD, Lee BE, Acosta N, Bautista M, Hubert CRJ, Hrudey SE, Frankowski K, Pang X-L (2024). Wastewater-based surveillance as a tool for public health action: SARS-CoV-2 and beyond. Clin Microbiol Rev. 37(1):e0010322. doi: 10.1128/cmr.00103-22.
- Paul JR, Trask JD, Gard S (1940). II. Poliomyelitic virus in urban sewage. J Exp Med. 71(6):765-77. doi: 10.1084/jem.71.6.765.
- Schilmann A, Sánchez-Pájaro A, Ovilla-Muñoz M, Téllez-Sosa J, Bravo-Romero S, Bahena-Reyes SY, Lobato M, Martínez-Barnetche J, Alpuche-Aranda CM, Lamadrid-Figueroa H, Barrientos-Gutiérrez T (2023). SARS-CoV-2 wastewater surveillance in ten Cities from Mexico. Water 15(4):799. doi: 10.3390/w15040799.
- Shephard DA (1989). John Snow and research. Can J Anaesth. 36(2):224-241. doi: 10.1007/BF03011450.
- Wilson WJ (1933). Isolation of enteric bacilli from sewage and water and its bearing on epidemiology. Br Med J. 2(3794):560-562. doi: 10.1136/bmj.2.3794.560.
- World Health Organization (WHO). WHO Director-General's opening remarks at the media briefing – 5 May 2023. 2023. Consultada el 24 de febrero de 2025 de https://www.who.int/news-room/speeches/item/who-director-general-s-opening-remarks-at-the-media-briefing---5-may-2023
- Xu X, Deng Y, Ding J, Zheng X, Wang C, Wang D, Liu L, Gu H, Peiris M, Poon LLM, Zhang T (2023). Wastewater genomic sequencing for SARS-CoV-2 variants surveillance in wastewater-based epidemiology applications. Water Res. 244:120444. doi: 10.1016/j.watres.2023.120444.
- Zarza E, Diego-García E, García LV, Castro R, Mejía G, Herrera D, Cuevas R, Palomeque Á, Iša P, Guillén K (2022). Monitoring SARS-CoV-2 in the wastewater and rivers of Tapachula, a migratory hub in southern Mexico. Food Environ Virol. 14(2):199-211. doi: 10.1007/s12560-022-09523-2.
Esta columna se prepara y edita semana con semana, en conjunto con investigadores morelenses convencidos del valor del conocimiento científico para el desarrollo social y económico de Morelos.









 Desarrollado por Linabat
Desarrollado por Linabat